"Son los encargados de reciclar las proteínas, pero si cumplen mal su cometido, por exceso o por defecto, aparecen diversas enfermedades habituales".
Es ampliamente conocida la gran diversidad de funciones que cumplen las proteínas en los seres vivos. Los tipos de proteínas que una célula produce dependen de los genes que se hallen activos en cualquier momento. Son éstos los que determinan la secuencia en que deben unirse las 20 subunidades básicas, los aminoácidos. Las cadenas luego se pliegan en hélices compactas para constituir diferentes clases de proteínas, dotadas cada una de una función específica que le viene impuesta por su forma y su composición química.
¿Qué ocurre cuando deja de necesitarse la presencia de esa proteína? Durante años se dio por supuesto que la degradación de las proteínas se realizaba en los lisosomas, sacos de enzimas digestivas habituales en la mayoría de las células del organismo. Pero a comienzos de los años setenta Goldberg demostró que las bacterias y los hematíes inmaduros, células privadas de lisosomas, podían, no obstante, destruir proteínas anómalas. Y más aún, el proceso requería energía, lo que no acontecía con otros procesos de degradación.
El equipo de Golberg consiguió que ese proceso de degradación dependiente de energía funcionase en el tubo de ensayo. Merced a ello, varios grupos de investigación descubrirían, a finales de los años setenta y a lo largo del decenio siguiente, las enzimas responsables. Por último, en 1988, dos grupos -dirigido uno por Goldberg y el otro por Martín C. Rechsteiner, de la Universidad de UTA- observaron que de la degradación de las proteínas se encargaban grandes complejos multienzimáticos. A estas macroestructuras el grupo de Goldberg les dio el nombre de proteosomas.
Se les llamó así porque contienen muchas proteasas, enzimas que degradan las proteínas. Pero los proteosomas centuplican el tamaño de otras proteasas y revisten mayor complejidad. Una vez colocada una proteína en el umbral de un proteosoma, penetra en el interior de la partícula y allí se descompone en los aminoácidos que la constituían, piezas que podrán luego aprovecharse para la síntesis de nuevas proteínas. La mayoría de las proteínas se renuevan al cabo de unos días, incluso en las células que raramente se dividen, como hepatocitos o neuronas. Según de qué proteína hablemos, así será su ritmo de degradación: unas tienen periodos de semivida de sólo 20 minutos, mientras que otras, en la misma célula, pueden subsistir días o semanas. Pero esas tasas de degradación varían de forma drástica según se modifiquen las condiciones de nuestro cuerpo.
A primera vista, la destrucción continua de los constituyentes de la célula parece un derroche; presta, sin embargo, un servicio inestimable a numerosas funciones esenciales. La degradación de una enzima importante o una proteína reguladora, por ejemplo, es un mecanismo habitual que las células utilizan para frenar o suspender una reacción química. Por otra parte, muchos procesos celulares se activan con la degradación de una proteína inhibidora clave. Esta eliminación de proteínas reguladoras reviste particular interés en la temporización de las transiciones entre las etapas del ciclo de la división celular.
La degradación de proteínas desempeña un papel crucial en la regulación general del metabolismo corporal. Por ejemplo, en el estado de desnutrición o de enfermedad, la vía de los proteosomas se activa en nuestros músculos, proporcionando aminoácidos que pueden convertirse en glucosa requerida para la combustión energética. Se refleja esa degradación excesiva de proteínas en el desgaste y debilidad muscular de los desnutridos y en los pacientes con cáncer avanzado, sidosos y diabéticos sin tratar.
Nuestro sistema inmunitario en su constante búsqueda de células infectadas con virus o cancerosas para eliminar, depende también de los proteosomas para generar las señales que identifiquen a tales células dañinas. En efecto, aunque suelen degradarse las proteínas celulares hasta dejarlas en sus aminoácidos, los proteosomas dejan sueltos unos pocos fragmentos de entre ocho y diez aminoácidos, que son capturados y presentados en la superficie de la células, donde el sistema inmunitario comprueba si son normales o anormales. En estados patológicos y en el bazo y los ganglios linfáticos se producen inmunoproteosomas, tipos de proteosomas especializados que potencian la eficacia de este mecanismo de vigilancia.
Gracias a la degradación de las proteínas por los proteosomas se evita la acumulación de proteínas aberrantes, potencialmente tóxicas. Las células de mamíferos y las bacterianas destruyen de una manera selectiva las proteínas que muestran conformaciones anómalas causadas por una mutación, por errores en la síntesis o por alteraciones de otra clase.
En múltiples enfermedades genéticas humanas se deja sentir la importancia de la degradación de proteínas anómalas. En varias anemias hereditarias, un gen mutante determina la síntesis de moléculas anormales de hemoglobina, que no se pliegan adecuadamente y las destruyen los proteosomas inmediatamente después de la síntesis. Por su parte, la fibrosis quística está causada por una mutación en el gen que cifra una proteína en forma de poro, que traslada el cloruro a través de la membrana externa de la célula. Debido a que estos mutantes de los transportadores de cloruro desarrollan a menudo una conformación inadecuada, los proteosomas los degradan antes de que alcancen la membrana celular. El moco pegajoso que se acumula en los pulmones y en otros órganos de las personas con fibrosis quística obedece a la falta de transportadores de cloruro normales.
Otras enfermedades podrían ser, en parte, resultado de un fracaso de los proteosomas en su función degradadora de proteínas anómalas. Hay conjuntos de proteínas mal plegadas que se acumulan junto con proteosomas en determinadas neuronas del cerebro de sujetos con la enfermedad de Parkinson, la de Huntington o la de Alzheimer. Averiguar por qué las neuronas de individuos afectados con estos trastornos neurodegenerativos dejan de degradar las proteínas anormales constituye hoy un campo de intensa investigación.
Arquitectura de los proteosomas
Desde la perspectiva de la proteína, los proteosomas son estructuras gigantes. Mientras que una proteína de tamaño medio tiene entre 40.000 y 80.000 daltons, la mayoría de los proteosomas de los organismos superiores pesan alrededor de millones de daltons. A mediado de los años noventa, un grupo dirigido por Wolfgang Baumeister y Robert Huber, del Instituto Max Planck de Bioquímica, recurrió al microscopio electrónico y a la técnica de la difracción de rayos X para determinar la arquitectura molecular de los proteosomas. Cada uno de ellos está constituido por una partícula central con aspecto de túnel a la que acompañan una o dos partículas reguladoras menores, situadas en un extremo o en ambos. La partícula central consta de cuatro anillos apilados -cada uno compuesto a sus vez por siete subunidades- que rodean un canal central, que constituye el tracto digestivo del proteosoma. Los dos anillos externos actúan como puertas de control y alejan así el peligro de que alguna proteína extraviada penetre en la cámara de destrucción.
A su vez, las partículas de los extremos se comportan como guardianes exigentes del paso hacia la partícula central. Son partículas reguladoras que reconocen y se unen a proteínas marcadas para la destrucción; luego, recurren a la energía necesaria para desplegar las proteínas y arrojarlas hacia la partícula central, donde se rompen en fragmentos de tamaño variable.
Se han sintetizado y aislado compuestos que inhiben de una manera selectiva el proteosoma sin afectar otras enzimas celulares, lo que podría comportar efectos secundarios. A través de tales inhibidores se ha venido revelando la complejidad de la vía proteosómica. A dosis elevadas, los inhibidores acaban produciendo la muerte de las células, lo que no debe sorprendernos si consideramos las importantes funciones desempeñadas por los proteosomas. No podemos omitir que, in vivo o in vitro, las células cancerosas parecen más sensibles a esos efectos letales que las células normales.
El momento de la muerte
El proteosoma no elige proteínas al azar para destruirlas. Corresponde a la célula señalar las proteínas destinadas a este fin. En su inmensa mayoría, esas proteínas se etiquetan primero con ubiquitina, que debe su nombre a que se encuentra en muchos organismos diferentes. Con sólo 76 aminoácidos, la ubiquitina es una proteína bastante pequeña; se une a proteínas mayores formando largas cadenas. Estas colas de poliubiquitina actúan como códigos postales que encaminan y aceleran las proteínas selladas hacia el proteosoma.
El control del momento de la muerte de una proteína no reside en la propia destrucción en el proteosoma, sino en el proceso de la incorporación de las cadenas de ubiquitina, la ubiquitinación, que requiere energía.
El proceso de ubiquitinación de las proteínas se desarrolla en varias etapas. En él participan tres enzimas: E1, E2 y E3. La enzima E1 activa la ubiquitina y la asocia con E2. La tercera enzima, E3, facilita entonces la transferencia de la ubiquitina activada desde E2 hasta la proteína. El proceso se repite hasta que una larga cola de ubiquitinas cuelga de la proteína. El proteosoma reconoce en ese instante la cadena en cuestión y atrae la proteína hacia su interior.
Para conocer por qué se elige una determinada proteína para la ubiquitinación hay que profundizar en las enzimas E3. Se ha descubierto que existen centenares de E3 distintas que reconocen la información encerrada en las secuencias de aminoácidos de otras proteínas que las convierten en objetivo de la ubiquitinación. Ante condiciones fisiológicas alteradas, pensemos en una infección o una falta de nutrientes, las células pueden modificar las proteínas mediante la adición de grupos fosfato. Esa fosforilación puede trastornar la actividad de una proteína o su capacidad para unirse a las E3. Estas enzimas reconocen también las proteínas que fracasan en el plegamiento o que están alteradas; las hacen entrar en el proceso de limpieza después de marcarlas para que el proteosoma las aprehenda. Muchos procesos celulares clave se apoyan en la estabilidad de las proteínas; por tanto , si se descifra cómo se controla la estabilidad, se tendrá la llave de numerosos secretos de la biología.
Mediante el control de la estabilidad de proteínas cruciales, las enzimas E3 regulan el desarrollo de las extremidades, la respuesta inmunitaria, la división celular y la comunicación intercelular, entre otras. Los propios ritmos circadianos y la floración de las plantas caen en la órbita de las enzimas E3. De varias proteínas E3 se conoce incluso su función supresora de tumores u oncogénica, lo que vincula la ubiquitinación al comienzo del proceso canceroso.
Así acontece con el supresor de tumores Von Hippel Lindau (VHL), una E3 que sufre a menudo una mutación en los tumores renales. La función de VHL consiste en demorar el crecimiento celular limitando el desarrollo de los vasos celulares en los tejidos; cuando se produce la mutación, los tumores recién formados generan un rico suministro sanguíneo y crecen rápidamente. Se acaba de encontrar que cierta forma hereditaria de la enfermedad de Parkinson se origina en virtud de una mutación del gen de un tipo de la enzima E3, inductora de la acumulación de proteínas en ciertas células cerebrales y de su aniquilamiento.
Los virus, que se distinguen por derivar en su propio beneficio los procesos celulares, han evolucionado hasta adquirir medios para secuestrar el mecanismo de ubiquitinación y degradación de proteínas en sus intereses. Sirven de ejemplo los virus del papiloma humano (VPH), que provocan verrugas genitales, cáncer de útero o cáncer de recto. La transformación hacia el desarrollo tumoral se bloquea, habitualmente, por la p53, una de las proteínas supresoras de tumores en el organismo. El VPH recurre a una estratagema para evitar el sistema defensivo celular: fabrica una proteína que se enlaza simultáneamente a la p53 y a una enzima E3. La fatídica unión insta la ubiquitinación de la p53, que así queda marcada para su destino fatal en el proteosoma. Las células, indefensas, se convierten más fácilmente entonces en un cáncer incipiente.
De un truco similar se vale el VIH para destruir la CD4 de la superficie celular, proteína que necesita el virus para infectar las células, aunque bloquea la producción de más virus en una fase ulterior. CD4 constituye el sitio que le permite al VIH penetrar en las células T del sistema inmunitario; se une a la proteína gp160 que sobresale de la superficie del virus. Pero cuando el VIH se apresta a replicarse en las células recién infectadas, la proteína CD4 puede constituir un problema: se adhiere a las proteínas gp160 recién sintetizadas, evitando así que se reúnan con otras proteínas víricas y formar nuevos virus. Para obviar tal obstáculo, el VIH ha desarrollado la Vpu, una proteína que se une a la CD4 y a un complejo en el que se integra una enzima E3, lo que hace que la proteína CD4 se ubiquitina y caiga después en el proteosoma destructor.
Están apareciendo nuevos datos que realzan la importancia de las E3. No cabe duda de que estas enzimas atraerán el desarrollo de fármacos en el futuro. Puesto que cada E3 es responsable de la destrucción de un número restringido de proteínas, los inhibidores de las E3 deberían ser fármacos muy específicos con pocos efectos secundarios. La identificación reciente de grandes familias de enzimas E3 ha abierto nuevos caminos para la creación de medicinas. De esos hallazgos apasionantes se espera un mejor conocimiento de los fenómenos reguladores y de la propia biología humana. Cuanto más sepamos sobre los proteosomas y la maquinaria de selección para la ubiquitinación, tanto mejor apreciaremos cuánto debe la vida a la muerte de las proteínas.
El sistema inmunitario
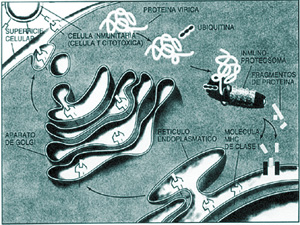
El sistema inmunitario se apoya en los inmunoproteosomas. Estos proteosomas especializados le ayudan a distinguir entre células sanas y células cancerosas o infectadas por virus. En el ejemplo de la imagen, una proteína vírica está marcada con ubiquitina para su destrucción por el inmunoproteosoma. Trozos de la proteína vírica, que constan de ocho a diez aminoácidos, entran entonces en el retículo endoplasmático, donde se unen a moléculas recién sintetizadas, que forman el complejo principal de histocompatibilidad (MHC) de clase l. A medida que las moléculas MHC de clase I cursan por el aparato de Golgi y salen a la superficie celular, van captando los fragmentos de las proteínas víricas. Las células T citotóxicas, del sistema inmunitario, reconocen como material foráneo los trozos del virus encastrados en las moléculas MHC de clase I en la superficie celular y destruyen la célula infectada.